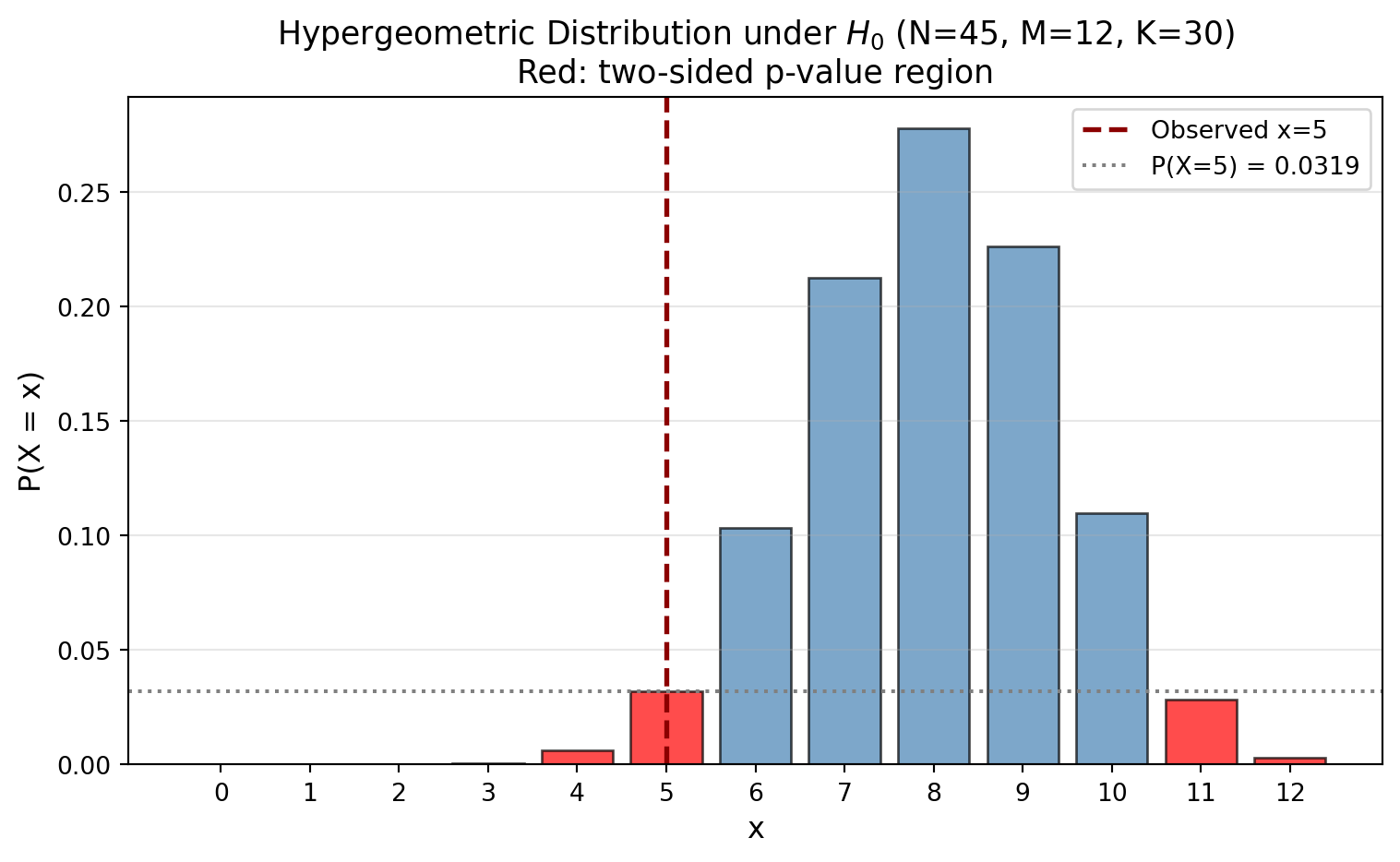
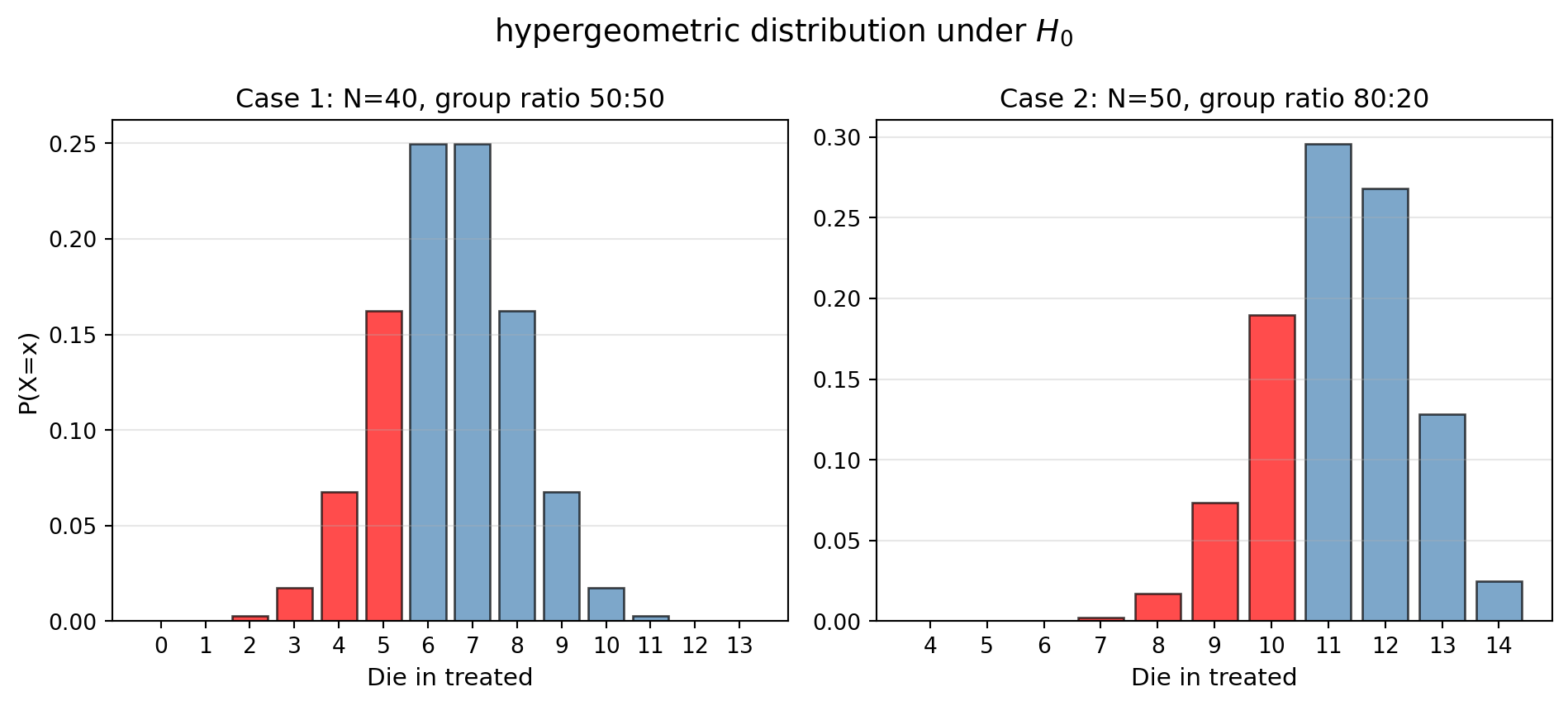
from scipy import stats
from typing import Literal, Iterable
from multiprocessing import Pool, cpu_count
def _power_contribution(
r_values: Iterable[int],
n1: int,
n2: int,
p1: float,
p2: float,
alpha: float,
alternative: str,
) -> float:
"""r の集合に対する検出力寄与を計算"""
power = 0.0
# Binomial PMF を事前計算
binom1 = stats.binom(n1, p1)
binom2 = stats.binom(n2, p2)
for r in r_values:
x_min = max(0, r - n2)
x_max = min(n1, r)
for x in range(x_min, x_max + 1):
table = [[x, n1 - x], [r - x, n2 - (r - x)]]
_, p_value = stats.fisher_exact(table, alternative=alternative)
if p_value < alpha:
power += binom1.pmf(x) * binom2.pmf(r - x)
return power
def power_fisher_exact(
p1: float,
p2: float,
n1: int,
n2: int,
alpha: float = 0.05,
alternative: Literal["two-sided", "greater", "less"] = "two-sided",
multi: bool = False,
n_workers: int | None = None,
) -> float:
"""
Fisher正確検定の検出力を解析的に計算する。
Mainland and Sutcliffe (1953) の検出力関数に基づき、
Thomas and Conlon (1991) のアルゴリズムを使用して効率的に計算します。
検出力は以下の式で計算されます:
β(n1, p1, p2) = Σ_{r=0}^{2n} Σ_{x∈C_r} C(n,x) * C(n,r-x) *
p1^x * (1-p1)^{n-x} * p2^{r-x} * (1-p2)^{n-r+x}
ここで、rは両群の成功数の合計、xは第1群の成功数、
C_rは超幾何分布による臨界領域です。
Parameters
----------
p1 : float
第1群の真の比率 (0 < p1 < 1)
p2 : float
第2群の真の比率 (0 < p2 < 1)
n1 : int
第1群のサンプルサイズ
n2 : int
第2群のサンプルサイズ
alpha : float, optional
有意水準 (デフォルト: 0.05)
alternative : {"two-sided", "greater", "less"}, optional
対立仮説の種類 (デフォルト: "two-sided")
multi : bool, optional
マルチプロセス並列化を使用するかどうか (デフォルト: False)
n_workers : int or None, optional
ワーカー数 (デフォルト: CPU数)。multi=Trueの場合のみ有効
Returns
-------
float
検出力 (0から1の間の値)
Examples
--------
>>> power_fisher_exact(0.5, 0.9, 20, 20)
0.7123 # 解析的に計算された検出力
>>> power_fisher_exact(0.5, 0.9, 100, 100, multi=True)
0.9999 # マルチプロセスで高速化
Notes
-----
- シミュレーションと異なり、正確な値を返します
- 計算量は O(n1 * n2) であり、大きなサンプルサイズでも高速です
- multi=Trueにすると複数CPUコアを使用して高速化できます
References
----------
- Mainland, D. and Sutcliffe, M.I. (1953). Statistical methods in
medical research. Canadian Journal of Medical Sciences, 31, 406-416.
- Thomas, R.G. and Conlon, M. (1991). Algorithm AS 259: Statistical
Algorithms. Applied Statistics, 40(1), 258-261.
"""
# 入力値の検証
if not (0 < p1 < 1):
raise ValueError(f"p1は0より大きく1より小さい値である必要があります: {p1}")
if not (0 < p2 < 1):
raise ValueError(f"p2は0より大きく1より小さい値である必要があります: {p2}")
if n1 <= 0:
raise ValueError(f"n1は正の整数である必要があります: {n1}")
if n2 <= 0:
raise ValueError(f"n2は正の整数である必要があります: {n2}")
if not (0 < alpha < 1):
raise ValueError(f"alphaは0より大きく1より小さい値である必要があります: {alpha}")
if alternative not in ["two-sided", "greater", "less"]:
raise ValueError(
f"alternativeは 'two-sided', 'greater', 'less' のいずれかである必要があります: {alternative}"
)
n = n1 + n2 # 総サンプルサイズ
M_values = list(range(n + 1))
# --- single process ---
if not multi:
return _power_contribution(
M_values, n1, n2, p1, p2, alpha, alternative
)
# --- multi process ---
if n_workers is None:
n_workers = cpu_count()
chunks = [M_values[i::n_workers] for i in range(n_workers)]
args = [
(chunk, n1, n2, p1, p2, alpha, alternative)
for chunk in chunks
if chunk
]
with Pool(n_workers) as pool:
results = pool.starmap(_power_contribution, args)
return sum(results)